了解等温滴定量热法 (ITC) 如何用于指导蛋白质 (RNA)/配体的结晶以及解决复合物结晶的常见问题。
Eric Ennifar, Cyrielle Da Veiga, Dominique Burnouf, Joelle Mezher, Barbara Puffer-Enders 和 Philippe Dumas.通讯作者:e.ennifar@unistra.fr Architecture et Réactivité de l’ARN, Institut de Biologie Moléculaire et Cellulaire, CNRS/Université de Strasbourg。
即使在表征良好的生物系统上,复合物(蛋白质/蛋白质、蛋白质/核酸、蛋白质/配体、核酸/核酸、核酸/配体)的结晶通常也非常枯燥无味,要么耗费时间,要么耗费样品。如果使用生物物理方法对复合物进行适当初步表征,可以显著提高复合物结晶的成功率。动态光散射 (DLS) 是评估样品结晶性的关键,这一点已得到充分证实 (1)。同样,已经开发出 DSC 基于差示扫描荧光(又称 ThermoFluor)的优化策略,可以促进蛋白质/配体的结晶 (2)。等温滴定量热法 (ITC) 是研究分子相互作用的“金标准”技术,我们在此表明,其可以成为一种有价值的技术来改善复合物的结晶。ITC 是一种真正的溶液中技术,在一次实验中直接提供两个分子之间完整的结合情况:两个分子之间的结合亲和力 (Ka)、焓变和熵变(ΔH 和 ΔS)以及化学计量学结果 (N) 都可以非常精确地获得 (3-5)。与其他类似生物物理方法相比,ITC 的一个主要优势是不受大分子尺寸上限或下限的限制,没有缓冲区限制。此外,最重要的在于对于结构研究而言,无需任何标记。此外,我们最近的研究表明,现代 ITC 设备和新的处理方法也允许在比通常想象的更多样化的系统上获得完整的动力学描述,从简单的配体结合到复杂的 RNA 折叠 (6)。
ITC 的主要限制是实验需要相对大量的样品。然而,这不应成为参与核磁共振或 X 射线晶体学研究的结构生物学家的瓶颈,因为这些分析的样品要求与 ITC 分析极为相似。由于样品在 ITC 实验中不会被损坏,因此可以回收并浓缩,用于后续的结晶生成实验。最后,ITC 还可用于评估核酸或复合物中使用的蛋白质是否正确折叠。我们将在此介绍这种 ITC 指导下结晶的示例。所有的实验均在 Microcal™ iTC200 (Malvern Instruments Ltd) 上进行。
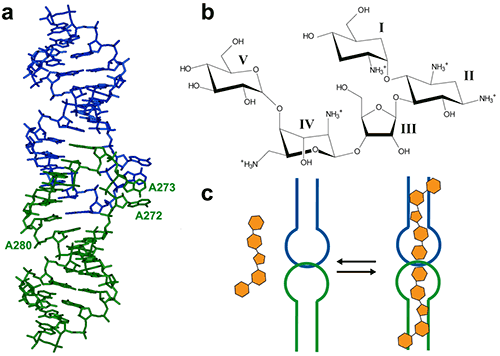
HIV-1 基因组 RNA 二聚作用起始位点 (DIS) 是一段高度保守的序列,具有茎环结构。该环状结构包含一段自身互补序列,通过在两个 DIS 发夹之间形成环-环(接吻环)复合物,促进病毒基因组的二聚作用。DIS 环-环复合物的晶体结构 (7) 显示出与细菌 16S 核糖体氨基酰-tRNA 位点(A 位点)的非预期相似性,后者是氨基糖苷类抗生素的作用靶点 (8)。因此,研究表明 HIV-1 的 DIS RNA 也可以与氨基糖苷结合,因此可以成为合理设计针对 HIV-1 基因组 RNA 的新药的极具吸引力的靶点 (9)。在此我们将展示 ITC 如何用来指导由氨基糖苷类药物利维霉素和 HIV-1 DIS RNA 吻环复合物组成的复合物的共结晶。
|
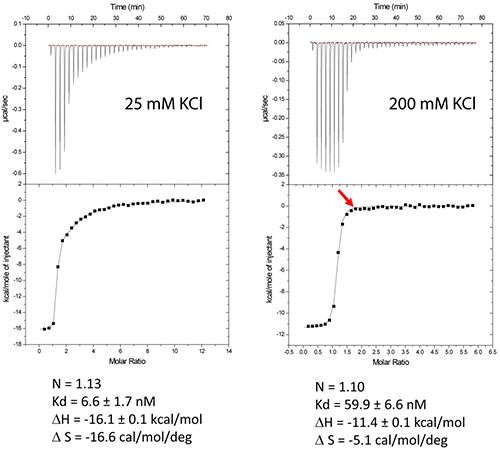
DIS/利维霉素相互作用提供了一个盐依赖性结合的示例,正如在核酸中经常观察到的情形。与许多核酸配体一样,氨基糖苷类药物携带正电,在低盐浓度下,由于静电相互作用而产生的非特异性相互作用占主导地位。然而,由于静电屏蔽作用,盐浓度的过度增加可能会阻止配体结合。因此,可以在不同盐条件下进行初步 ITC 实验以减少非特异性结合,同时保持特异性相互作用。在低盐缓冲液(25 mM KCl、2 mM MgCl2、25 mM 二甲基砷酸钠,pH 7.0)中进行的实验显示存在两个结合事件,不能用单一位点模型进行拟合(图 2A)。采用两个独立结合位点模型进行拟合,显示第一个结合非常紧密,与预期结果相似,具有 1:1 的化学计量学结果(每个茎环具有 1 个配体,吻环复合物具有 2 个配体)。而第二个结合位点的亲和力在较低微摩尔范围内,相当于非特异性相互作用。因此,这种非特异性结合可能阻碍在含有 >100 mM 浓度复合物的结晶条件下形成均匀 RNA/配体复合物。然而,在高盐条件下(200 mM KCl、2 mM MgCl2、25 mM 二甲基砷酸钠,pH 7.0)仅观察到特异性和紧密地结合(图 2B)。因此,在寻找结晶条件时,应首选这种导致均匀 RNA/配体复合物的实验条件。
错误折叠是 RNA 结构研究中经常出现的问题。在此获得了预期的化学计量学结果,因此这些 ITC 实验也评估了 RNA 是否正确折叠。此外,还评估了是否使用正确的抗生素浓度。
|
ITC 的另一个有趣的观点是能够实时监测化学计量学结果,以便在达到所需的 RNA/配体比例(应为 ≥1.0)时停止实验。例如在 200 mM KCl 的 DIS RNA/利维霉素滴定实验中,当用配体饱和 RNA 后,可以在第 12 次注射时停止实验(红色箭头)。应当在可以轻松估计化学计量学结果的情况下考虑该方法,即当产生的 S 型曲线斜率相当陡峭(Wieseman 系数 c ≥ 100)且解离速率常数 (koff) 应较缓慢。在此情况下,该方法可以避免样品中存在过量配体,并以最佳 RNA/配体复合物比例进行结晶。
下文使用一个标准示例,用非优化的盐浓度进行 DIS 吻环 RNA/利维霉素相互作用(ITC 缓冲液由 150mM KCl、5mM MgCl2、20mM 二甲基砷酸钠制成,pH 7.0)。所用的 RNA 序列是一段化学合成的长度为 23 个核苷酸的序列。HIV-1 亚型 A DIS 在第三位上含有一个溴尿核苷 (7),以便使用多波长反常散射 (MAD) 方法进行晶体结构测定。在 ITC 缓冲液中制备浓度为 120 μM 的 RNA 样品(以 RNA 链为单位),并置于 ITC 池中。注射器中注入 2.4mM 利维霉素的 ITC 缓冲液。在 12°C下进行 ITC 分析以改善复合物形成(因为相互作用为放热反应),并保护 RNA 免受可能的降解。
|
在 ITC 实验后,回收 ITC 池中形成的复合物,并使用微量浓缩器浓缩约三次(在 4°C 下进行以保存 RNA/配体相互作用)至约 400 µM。然后使用市售结晶稀疏基质筛选(crystallization sparse matrix screens,包含 96 种不同的结晶条件)和结晶自动化工作站将该溶液在结晶盘中混合(1/1 体积)。结晶库中充满了稀疏基质溶液,并将托盘置于 37°C 下进行孵育。使用 Hampton Research® Natrix HT 稀疏基质条件孵育 16-24 小时后(图 4),应生长出适合进行衍射(使用同步辐射 X 射线源最高可达到 1.6 Å 分辨率)和结构测定的大单晶(图 4)。
|
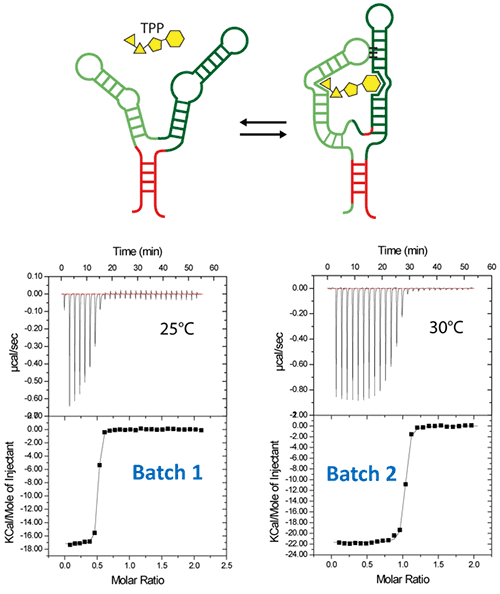
核糖开关是信使 RNA 的非翻译区,其可以与特定代谢物结合。结合后即可观察到构象转换,从而调节参与核糖开关底物的生物合成的蛋白质的表达 (12)。大肠杆菌 ThiC TPP 核糖开关对辅酶硫胺素焦磷酸 (TPP) 产生应答,而 TPP 是维生素 B1 的一种活性形式 (13) (图 5,顶部图)。我们在此示例中使用了长度为 85 个核苷酸的合成 RNA 片段,对应于 Ecoli TPP 适配体结构域 (6)。这种相当大的具有复杂二级和三级结构的 RNA 的一个主要问题是在超量生产、纯化和体外重新变性过程后如何恢复正确折叠的分子。
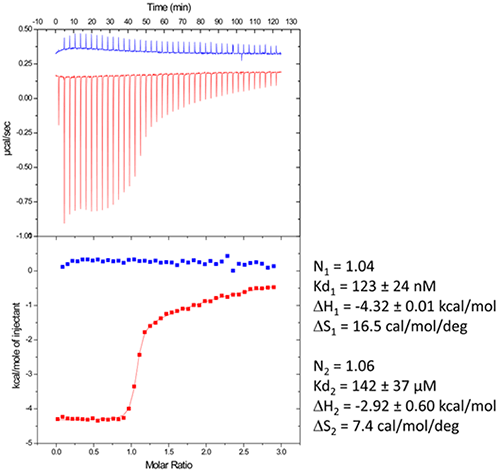
第一步,尝试了以下再折叠实验方案(RNA 批次 1):纯化后,RNA 被热解,在冰上快速冷却,并置于 ITC 缓冲液中,该缓冲液由 pH 6.5 的 50 mM 二甲基砷酸钠、100 mM 乙酸钾、5 mM乙酸镁制成。RNA 再折叠后,在 25°C 下进行 ITC 实验:将 30 µM RNA 置于 ITC 池中,注射器中注入 500 µM TPP。此 ITC 实验显示,仅大约一半 RNA 能够与 TPP 配体结合,这有力地说明 RNA 存在严重的折叠错误问题(图 5,中间图,见“批次 1”)。然后回收、浓缩在 ITC 池中形成的 RNA/TPP 复合物,并用于上述 HIV-1 DIS 吻环 RNA 的结晶试验。然而,在这些结晶试验中仅观察到清晰的液滴(图 5,左下图),与 RNA 的主要部分发生折叠错误的假设吻合。
第二步,改变了 RNA 折叠实验方案:在 H2O 中 90°C 热变性 3 分钟,随后将核糖开关冷却至室温 40 分钟。加入 10X ITC 缓冲液至最终浓度为 1X,并将混合液在室温下进一步孵育 40 分钟。在第二批 RNA 上进行 ITC 实验 (30 µM RNA, 500 µM TPP) 的结果表明,此 RNA 再折叠实验方案可以获得完全活性的核糖开关,因为 100% 的 RNA 能够与 TPP 配体发生结合(图 5,中间图,见“批次 2”)。在浓缩并滴加结晶液滴后,可以获得分辨率最高 5 Å 的大单晶衍射图(图 5,右下图)。上述结果明确了在建立结晶试验之前使用 ITC 作为复合物形成的质量控制的价值。
|

|
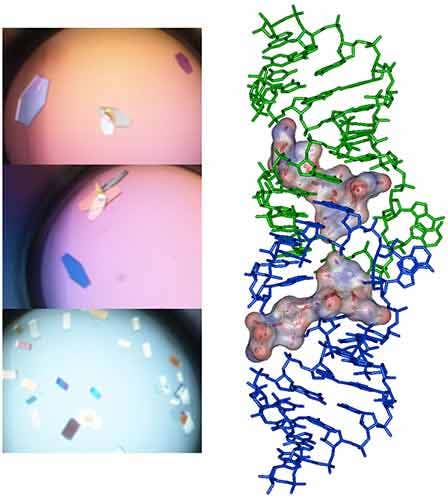
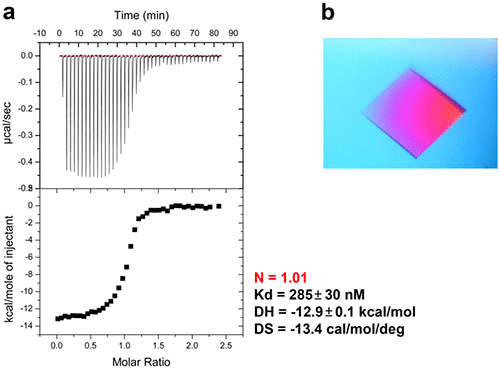
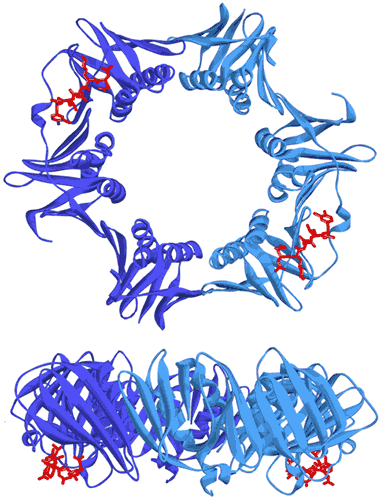
在细菌中,DNA 聚合酶持续合成因子(processivity factor,又称 β 滑动夹)是一个同源二聚体,它们通过一个保守小肽进入滑动夹疏水袋状结构而发生相互作用,从而赋予 DNA 聚合酶和其他参与 DNA 代谢的因子持续合成的能力 (14)。为了干扰这种相互作用,防止滑动夹与 DNA 聚合酶结合,已经开发了合成短肽以发挥抗生素作用 (15)。本研究旨在将与合成肽结合的铜绿假单胞菌滑动夹进行共结晶,获得对这种相互作用的结构性见解,并帮助开展基于结构的合理设计以获得具有更好亲和力的新抗生素肽。结晶前,进行 ITC 实验,将 β 滑动夹(35 µM,溶于 pH 7.4 的 10 mM Hepes、150 mM NaCl、3 mM EDTA 中)置于 ITC 池中,将肽 (300 µM) 注入注射器(图 6a)。该复合物在使用微浓缩器浓缩至 47 mg/mL 后进一步用于结晶试验。通过将 1 个体积的蛋白质/肽复合物与 1 个体积的储备液(0.1 M,pH 6.0,由 100 mM Mes 钠、100 mM CaCl2 和 20-30% [w/v] 聚乙二醇 [PEG] 400 制备)混合来进行结晶。在 20°C 下培养数日后,使用同步辐射获得了大的单晶衍射结果,最高分辨率为 2.2 Å(图 6b),并解析了 X 射线结构(图 7)。
|
|
可以在生物大分子复合物的结晶工作流程中应用 ITC 技术,并能作为提高结晶成功率的指导技术:
用于结构研究的样品要求非常适合进行 ITC 分析
评估真实的活性蛋白质和/或配体浓度(由化学计量学观测结果提供)
通过监测化学计量学结果,实时确定用于复合物结晶的最佳蛋白质/配体比例
帮助优化复合物形成(样品均匀性、亲和性、特异性)
可以“免费”提供相互作用的完整热力学特征。仅需在 ITC 分析后浓缩样品。
Ferre-D'Amare, A. R., and Burley, S. K. (1994) Use of dynamic light scattering to assess crystallizability of macromolecules and macromolecular assemblies, Structure 2, 357-359.
Dupeux, F., Rower, M., Seroul, G., Blot, D., and Marquez, J. A. (2011) A thermal stability assay can help to estimate the crystallization likelihood of biological samples, Acta Crystallogr D Biol Crystallogr 67, 915-919.
Ladbury, J. E., and Chowdhry, B. Z. (1996) Sensing the heat: the application of isothermal titration calorimetry to thermodynamic studies of biomolecular interactions, Chem Biol 3, 791-801.
Leavitt, S., and Freire, E. (2001) Direct measurement of protein binding energetics by isothermal titration calorimetry, Curr Opin Struct Biol 11, 560-566.
Privalov, P. L., and Dragan, A. I. (2007) Microcalorimetry of biological macromolecules, Biophys Chem 126, 16-24.
Burnouf, D., Ennifar, E., Guedich, S., Puffer, B., Hoffmann, G., Bec, G., Disdier, F., Baltzinger, M., and Dumas, P. (2012) kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry, J Am Chem Soc 134, 559-565.
Ennifar, E., Walter, P., Ehresmann, B., Ehresmann, C., and Dumas, P. (2001) Crystal structures of coaxially stacked kissing complexes of the HIV-1 RNA dimerization initiation site, Nat Struct Biol 8, 1064-1068.
Ennifar, E., Paillart, J. C., Marquet, R., Ehresmann, B., Ehresmann, C., Dumas, P., and Walter, P. (2003) HIV-1 RNA dimerization initiation site is structurally similar to the ribosomal A site and binds aminoglycoside antibiotics, J Biol Chem 278, 2723-2730.
Bernacchi, S., Freisz, S., Maechling, C., Spiess, B., Marquet, R., Dumas, P., and Ennifar, E. (2007) Aminoglycoside binding to the HIV-1 RNA dimerization initiation site: thermodynamics and effect on the kissing-loop to duplex conversion, Nucleic Acids Res.
Ennifar, E., and Dumas, P. (2006) Polymorphism of Bulged-out Residues in HIV-1 RNA DIS Kissing Complex and Structure Comparison with Solution Studies, J Mol Biol 356, 771-782.
Ennifar, E., Paillart, J. C., Bodlenner, A., Walter, P., Weibel, J. M., Aubertin, A. M., Pale, P., Dumas, P., and Marquet, R. (2006) Targeting the dimerization initiation site of HIV-1 RNA with aminoglycosides: from crystal to cell, Nucleic Acids Res 34, 2328-2339.
Winkler, W. C., and Breaker, R. R. (2005) Regulation of bacterial gene expression by riboswitches, Annu Rev Microbiol 59, 487-517.
Winkler, W., Nahvi, A., and Breaker, R. R. (2002) Thiamine derivatives bind messenger RNAs directly to regulate bacterial gene expression, Nature 419, 952-956.
Burnouf, D. Y., Olieric, V., Wagner, J., Fujii, S., Reinbolt, J., Fuchs, R. P., and Dumas, P. (2004) Structural and biochemical analysis of sliding clamp/ligand interactions suggest a competition between replicative and translesion DNA polymerases, J Mol Biol 335, 1187-1197.
Wolff, P., Olieric, V., Briand, J. P., Chaloin, O., Dejaegere, A., Dumas, P., Ennifar, E., Guichard, G., Wagner, J., and Burnouf, D. Y. (2011) Structure-based design of short peptide ligands binding onto the E. coli processivity ring, J Med Chem 54, 4627-4637.
Wolff, P., Amal, I., Olieric, V., Chaloin, O., Gygli, G., Ennifar, E., Lorber, B., Guichard, G., Wagner, J., Dejaegere, A., and Burnouf, D. Y. (2014) Differential modes of peptide binding onto replicative sliding clamps from various bacterial origins, J Med Chem 57, 7565-7576